基于其特异的结合特性,多年来DNA一直被当作骨架用来构造具有各种特殊光学或者电学性质的纳米材料。因此,如果能够对DNA链的三维结构有更好的认识,将极大地促进后续纳米材料的合成与制备。然而现有的技术并不能对DNA等具有柔性和特异性结构的微观物体进行有效的解析,无法直观地研究其结构-机理关系,从而极大地阻碍了相关研究的深入开展。
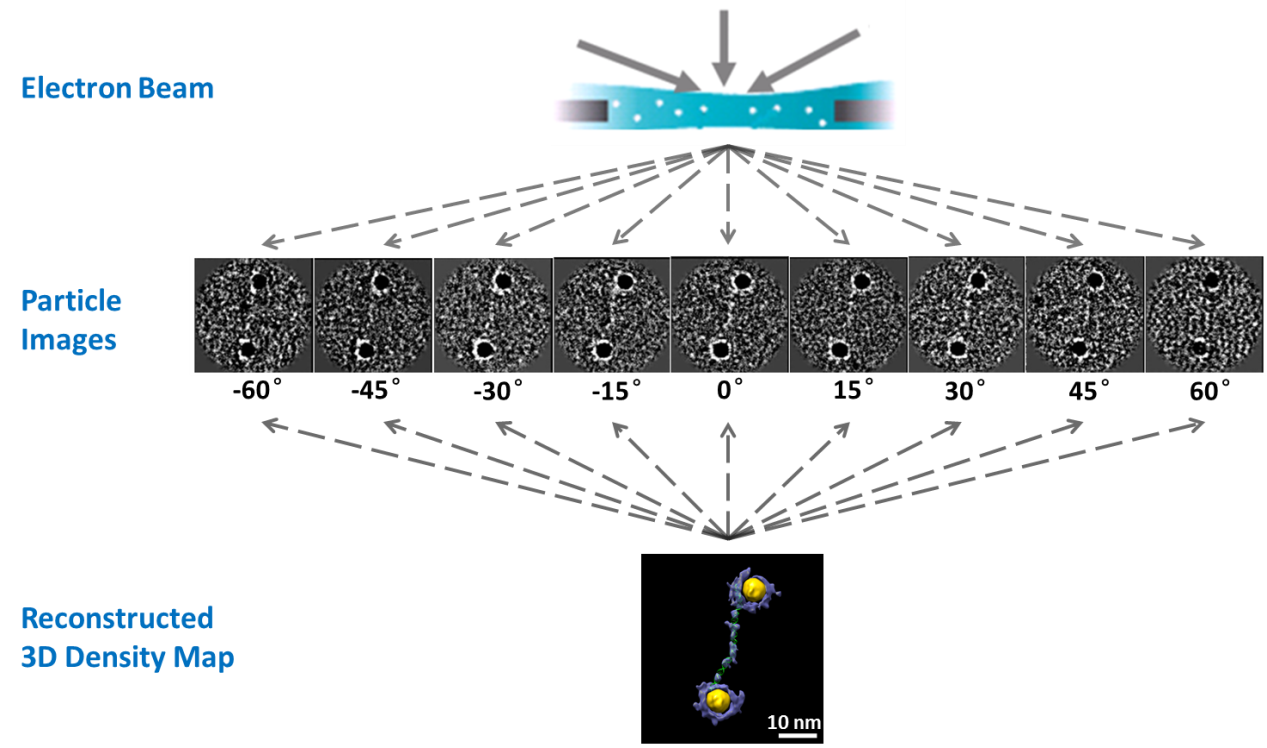
近日,理学院张磊教授、生命学院刘健康教授和美国劳伦斯伯克利国家实验室Gang Ren教授和Paul Alivisatos教授通过紧密的国际合作,利用各种透射电子显微镜技术和分子动力学模拟方法,得到了仅含有84个碱基对的单根双螺旋DNA的空间三维结构。该成果近日发表在国际综合学术刊物《自然通讯》上(Nature Communications, 影响因子11.470), 题目为“Three-dimensional structural dynamics and fluctuations of DNA-nanogold conjugates by individual-particle electron tomography”。张磊教授为第一作者,学院为第二作者单位,详见NPG网页:http://www.nature.com/ncomms/2016/160330/ncomms11083/full/ncomms11083.html
利用冷冻透射电镜技术,结合课题组开发的优化负染色技术和单分子电子断层成像(Individual-particle Electron Tomography,IPET)方法,此国际交叉联合科研组成功地得到了该DNA聚合物(含一个84碱基对的双螺旋DNA和两个5纳米的金颗粒)的高分辨冷冻电镜图像和负染色电子断层成像数据。基于这些数据,首次得到了14个此类聚合物的单分子电子密度图(分辨率约为1至2纳米);以这些电子密度图作为限制,利用分子动力学模拟手段,得到了14个双螺旋DNA的分子结构模型,并对其弹性模型进行了分析。结果表明,通过此方法得到的聚合物的形态统计分布和其它手段得到的结果一致,但是可以得到更多的结构信息,为进一步的机理分析提供了直接数据,对最终的复杂结构的设计、合成和验证提供了技术保障,也证明了课题组发展的单分子电子断层成像技术可以用于研究DNA复合物以及其他柔性蛋白质等有机大分子的动态结构。
张磊教授课题组长期从事软凝聚态物质大分子显微成像和结构与功能的研究,即应用先进的透射电子显微镜器材,发展和应用下一代软硬件技术,实现对单个软物质对象(尤其是结构活性高和分子量小的生物大分子,特别是具有极大生物医药学价值的蛋白质大分子)的纳米尺度的高分辨成像,并重构研究对象的高分辨三维结构,从而揭示其结构与功能等方面的研究。近期通过一系列的联合合作项目,与理学院张胜利教授、美国劳伦斯伯克利国家实验室Gang Ren博士、生命学院刘健康教授等科研团队展开了良好的合作,相继在Nature Chemical Biology,JACS,ACS Nano,Structure,Scientific Reports,J. Lipid Research,J. Biological Chemistry等国际著名高水平SCI期刊上发表20余篇论文。
该工作得到了西安交通大学“青年拔尖人才支持计划”,国家自然科学基金,科技部国家基础研究973项目、美国国立卫生院(NIH)和美国伯克利国家实验室Molecular Foundry的支持。